Visualization
Visualiseur de modélisation moléculaire gratuit et riche en fonctionnalités
Visualiser votre environnement scientifique
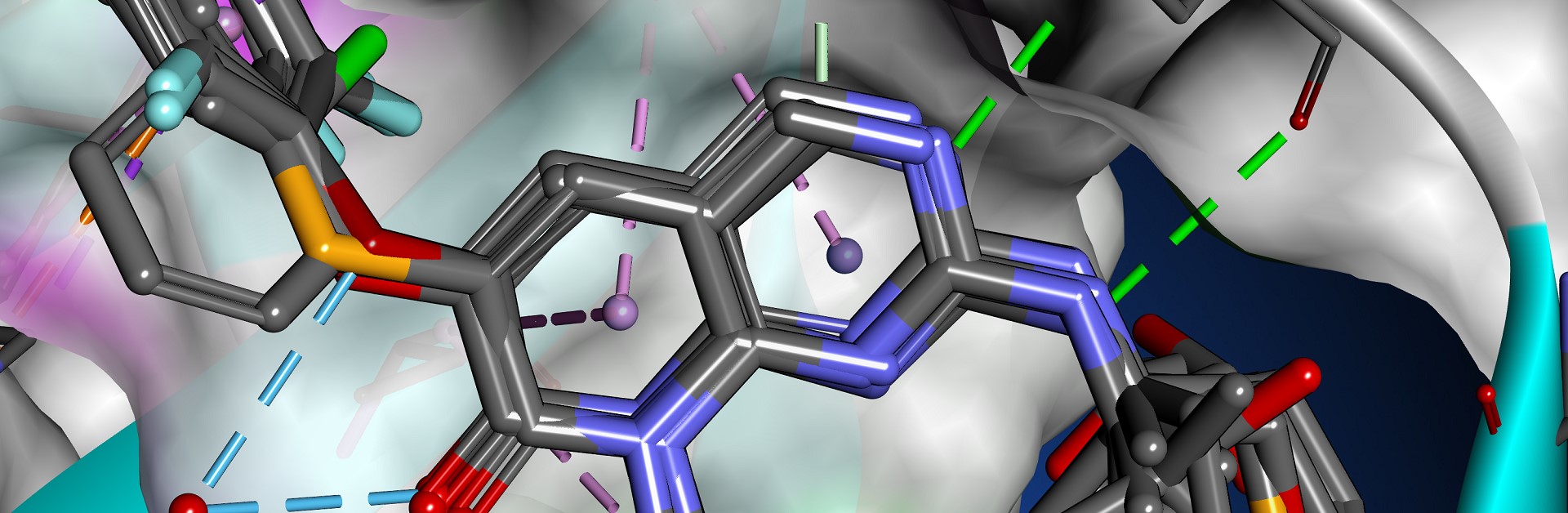
La visualisation des structures moléculaires est un aspect fondamental de l'analyse et de la communication des études de modélisation. Elle permet de mieux comprendre le mécanisme de la structure d'une molécule à visualiser. Les informations clés peuvent ainsi être partagées entre les experts en modélisation computationnelle et les autres membres de l'équipe concernés.
BIOVIA Discovery Studio Visualizer est une application de modélisation moléculaire gratuite et riche en fonctionnalités qui permet de visualiser, de partager et d'analyser les données sur les protéines et les petites molécules. Les experts et leurs collègues peuvent échanger des résultats de manière fluide et efficace, sans perdre de temps ni d'informations scientifiques.
Discovery Studio Visualizer est conçu pour saisir toutes les nuances de vos recherches :
- Graphiques de qualité supérieure, avec options d'affichage avancées et prise en charge stéréo
- Possibilité de générer des images de qualité de publication
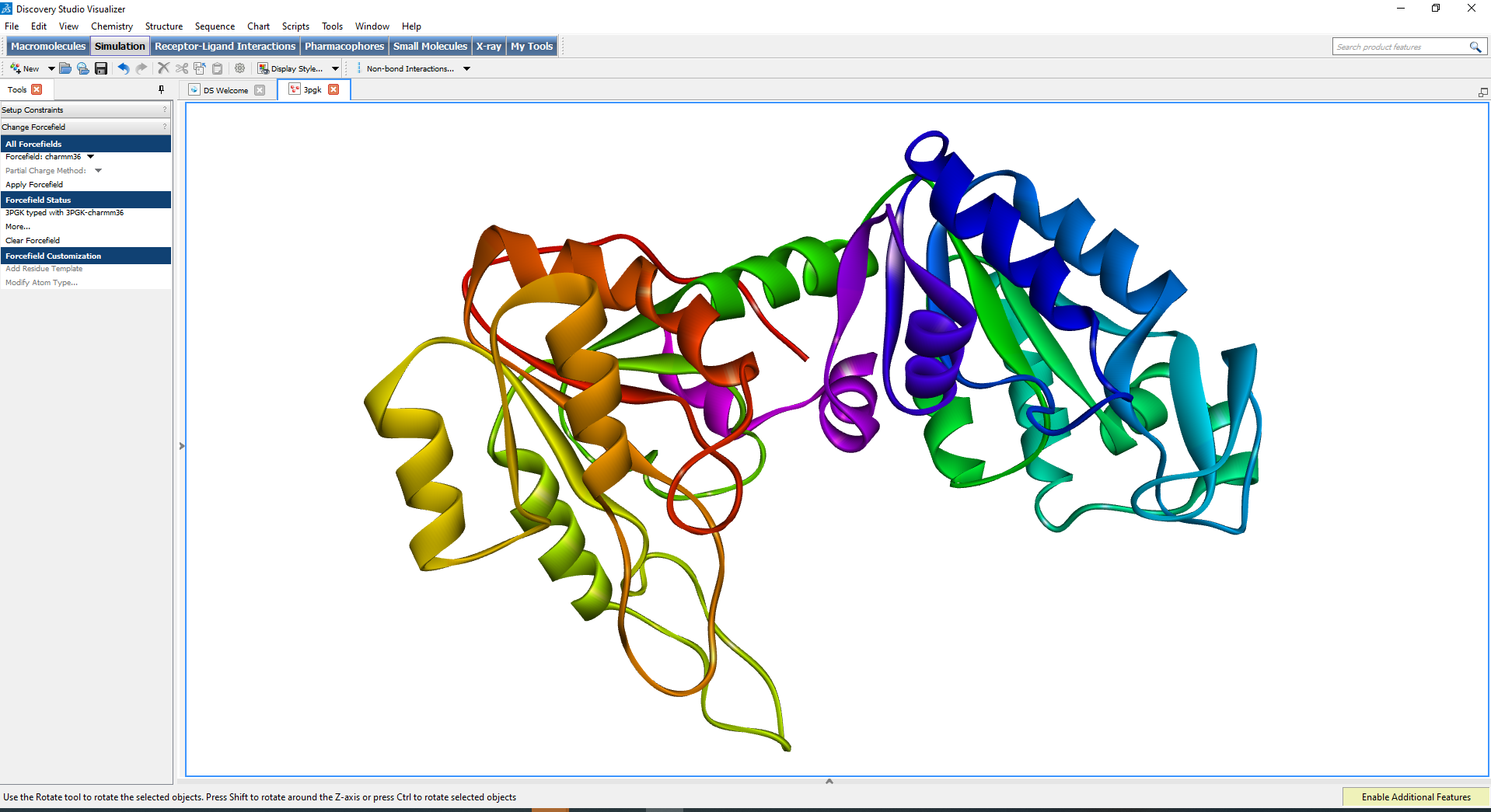
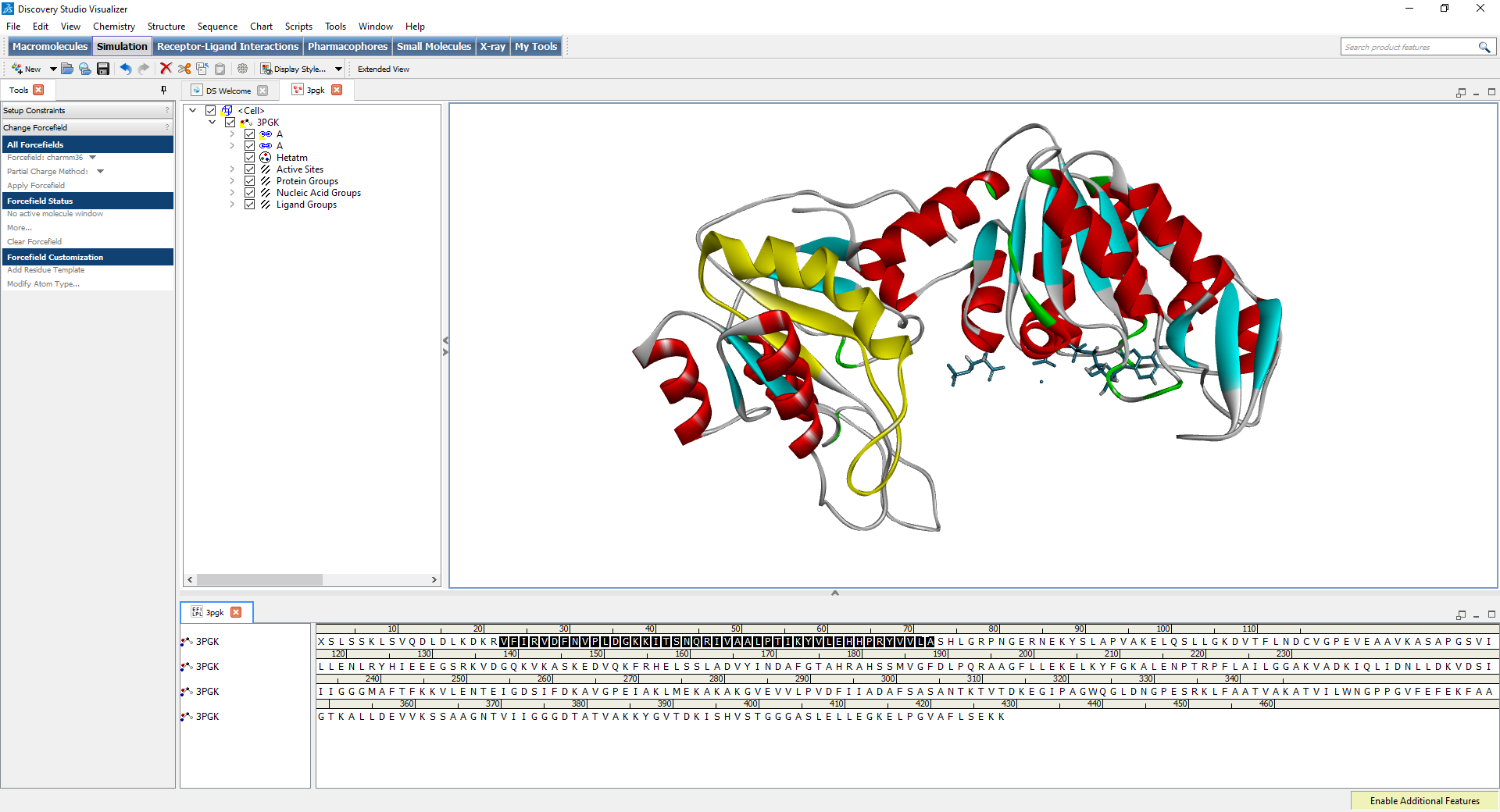
- Vue graphique 3D interactive assortie des vues arborescentes et de table de données associées
- Surfaces à plusieurs côtés et isosurfaces permettant d'améliorer la visualisation des structures moléculaires
- Possibilité de tracer des données en 3D, sous forme de lignes et de points et de graphiques à barres à partir de plusieurs séries de données
- Fonction de création de graphiques : cartes de chaleur, histogrammes, tracés de taux de réussite et bien plus encore
- Fonctionnalité de storyboard permettant de capturer une série de vues moléculaires à présenter et à partager
- Exportation des storyboards au format film
- Fonctionnalités de tri, de filtrage et de regroupement disponibles pour les propriétés dans la table de données
- Simplicité d'utilisation grâce aux scripts Perl permettant d'automatiser les tâches répétitives ou de lier des tâches entre elles
- Possibilité de personnaliser les panneaux et les barres d'outils, et la disposition des éléments de l'interface
- Prise en charge d'une grande variété de formats de fichiers de structures et de séquences
- Calculs RMS disponibles à différents niveaux de détail
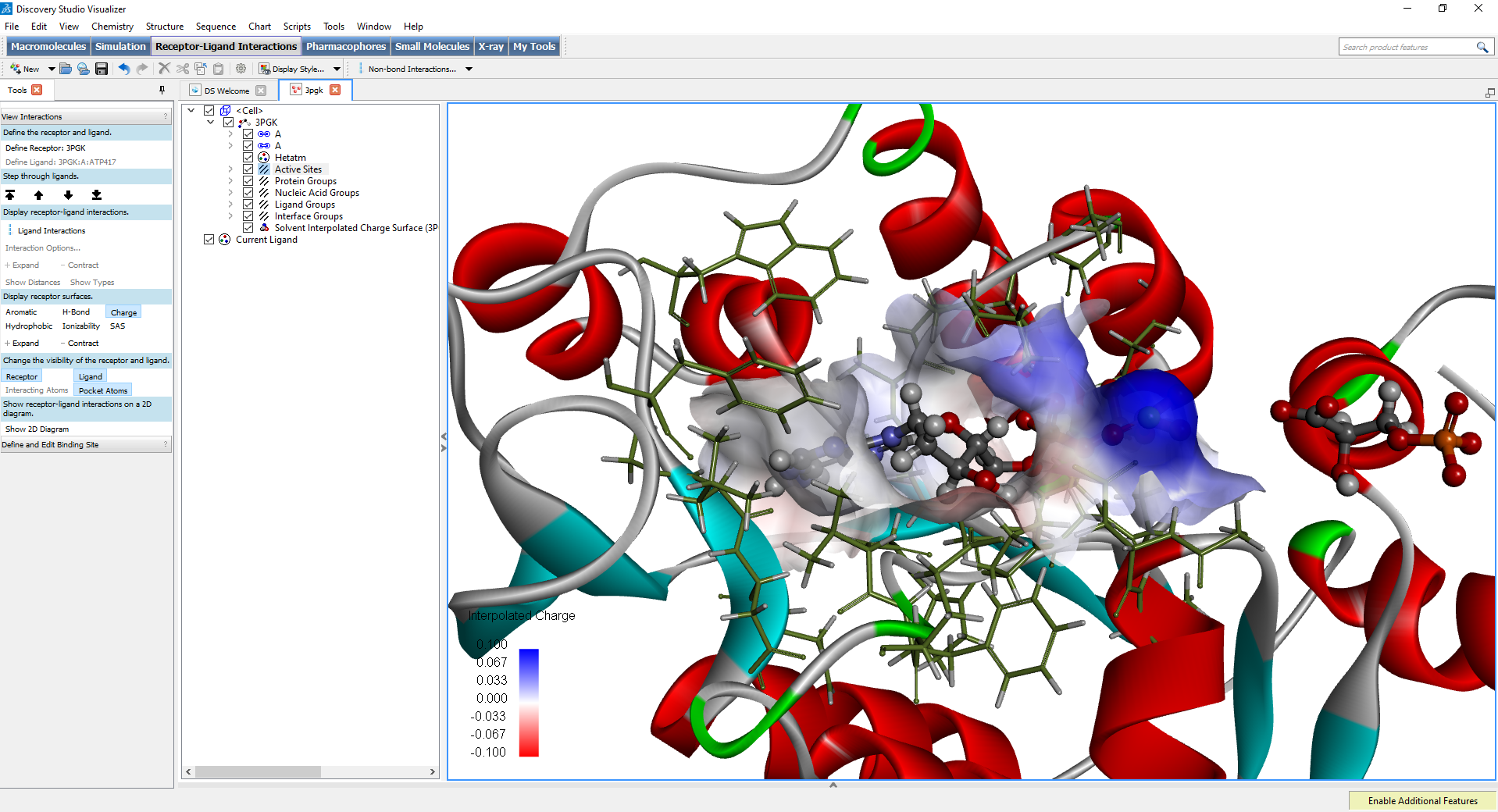
- Fonctions de surveillance des interactions non-liantes favorables, défavorables et non satisfaites
- Constructeurs moléculaires pour peptides et acides nucléiques
- Calcul de l'accessibilité aux solvants visant à identifier les résidus enfouis et exposés
- Fonctionnalité graphique, y compris les tracés de contact et le diagramme de Ramachandran
- Chargement direct des structures de protéines à partir de la base de données PDB
- Génération de rapports sur les protéines pour résumer les données des structures sous-jacentes
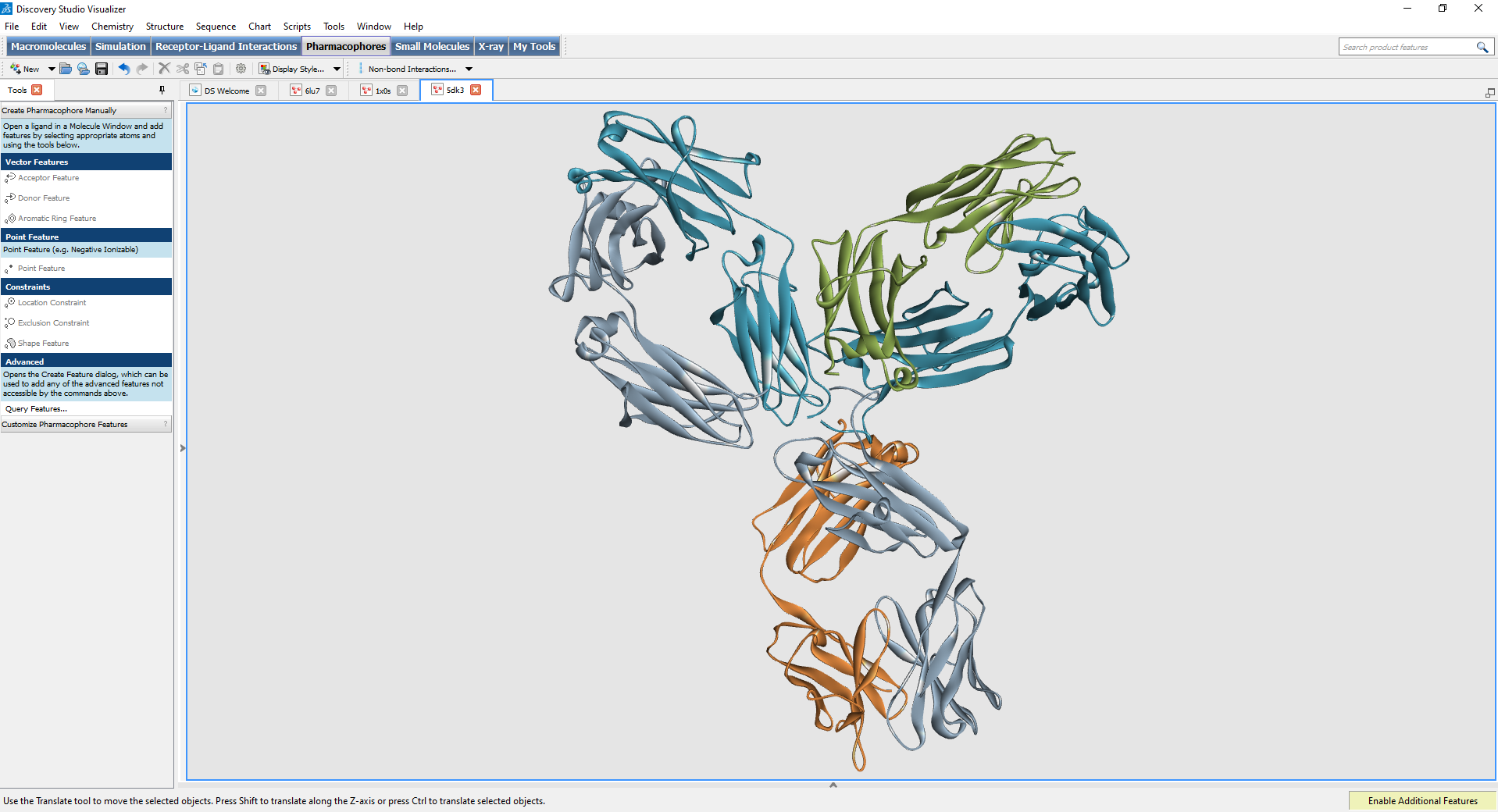
- Construction de l'unité biologiquement active d'une protéine à partir de ses sous-unités
- Fonctionnalité de nettoyage de protéines permettant d'ajouter des chaînes latérales manquantes, d'éliminer les désordres et de standardiser la dénomination des atomes
- Affichage amélioré des séquences de protéines et d'acides nucléiques ; analyse de leur composition et de leur alignement
- Superposition des structures en fonction des ancrages, des résidus et de l'alignement des séquences
- Prédiction de la structure secondaire des séquences de protéines
- Fenêtre d'annotation permettant de consulter et de modifier les annotations des séquences d'acides nucléiques et de protéines
- Affichage et traçage des contours de cartes de densité électroniques par rayons X
- Outils de base pour modifier les structures de rayons X
- Échantillonnage des conformations de chaînes latérales des rotamères et analyse des interactions
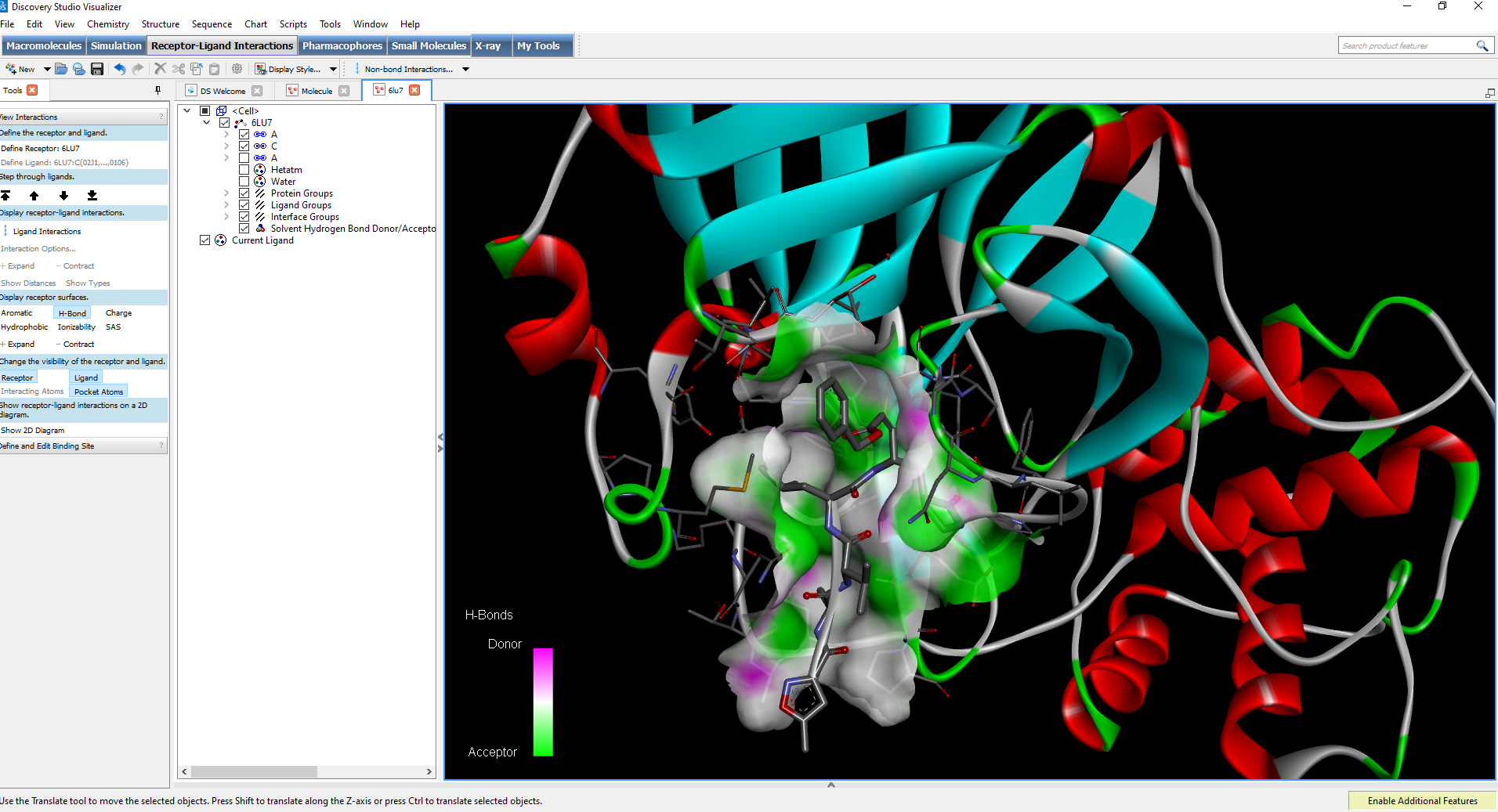
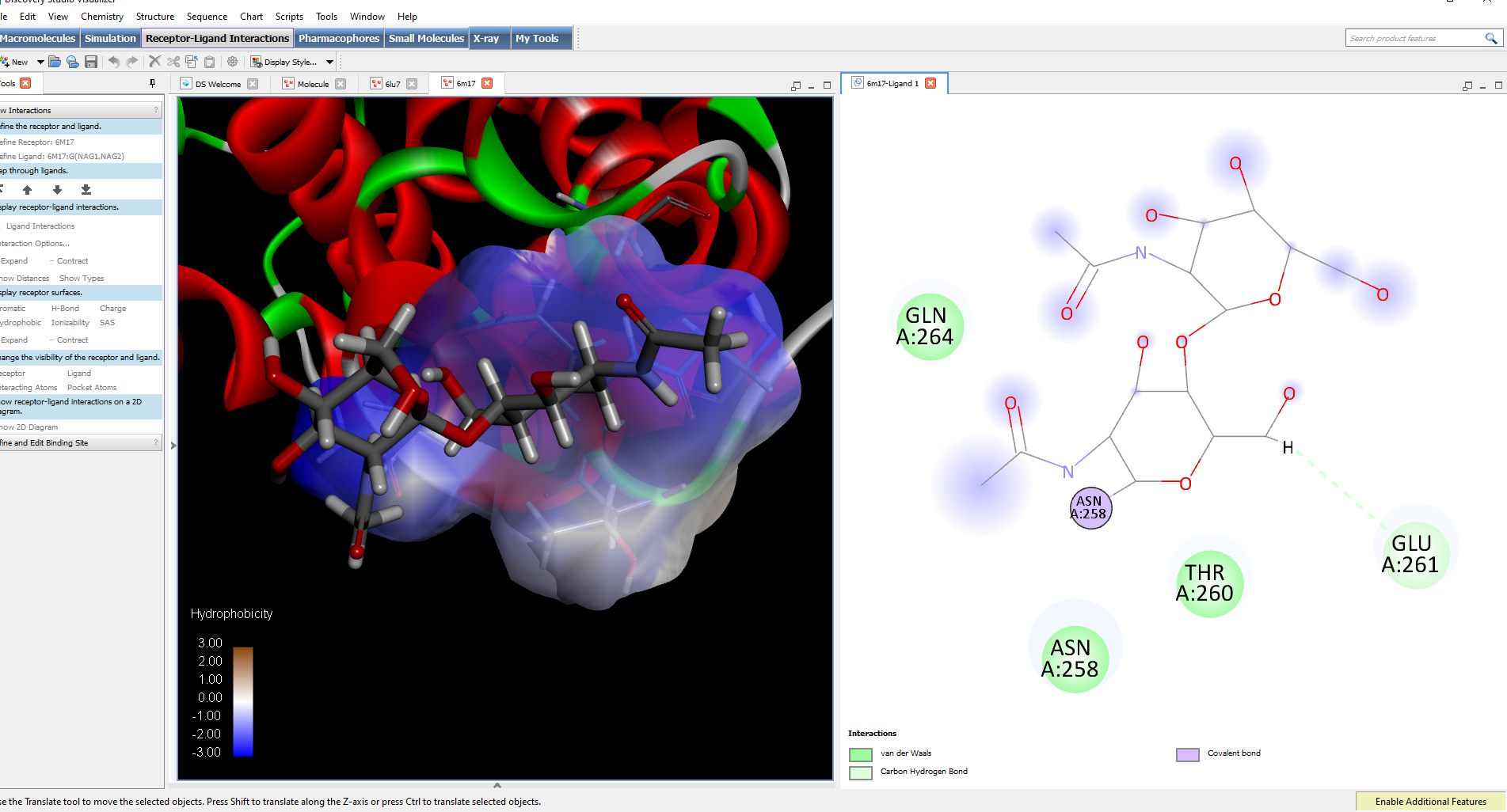
- Surfaces d'interaction récepteur-ligand affichant l'hydrophobicité, la liaison hydrogène, l'aromaticité et bien plus encore
- Affichage de la représentation 2D des molécules dans la table de données
- Outils d'esquisse permettant de créer des petites molécules
- Options de création et de modification de petites molécules 3D personnalisées à l'aide d'un panneau d'outils de fragments prédéfinis
- Optimisation de la géométrie des structures créées avec un champ de force rapide de type Dreiding
- Option de calcul des propriétés moléculaires de base (formule et masse, par ex.)
- Superposition des structures en fonction des molécules via l'alignement de champ ou des ancrages
- Définition, affichage et modification des sites de liaison de ligands
- Génération de tracés 2D des interactions récepteur-ligand
- Analyse des schémas de liaison entre une protéine et ses ligands
- Génération manuelle de requêtes sur les pharmacophores (Catalyst)
Commencer votre parcours
Le monde de la conception de médicaments évolue. Découvrez comment garder une longueur d'avance avec BIOVIA.
Rejoignez la conversation dans la communauté d'utilisateurs BIOVIA Drug Discovery & Development
À découvrir également
Découvrir comment BIOVIA peut vous aider
Contactez un expert BIOVIA pour découvrir comment nos solutions permettent une collaboration transparente et une innovation durable dans des entreprises de toutes tailles.
Commencer
Des formations et des cours sont disponibles pour les étudiants, le monde académique, les professionnels et les entreprises. Trouvez la formation BIOVIA qui vous correspond.
Obtenir de l'aide
Obtenez des informations sur la certification des logiciels et du matériel, les téléchargements de logiciels, la documentation utilisateur, les coordonnées du support et l'offre de services.